このブログでは、統計解析ソフトStataのプログラミングのTipsや便利コマンドを紹介しています.
Facebook groupでは、ちょっとした疑問や気づいたことなどを共有して貰うフォーラムになっています. ブログと合わせて個人の学習に役立てて貰えれば幸いです.
さて、本日の記事は、生存時間後のC-statiscicsを異なるモデルで算出したときに比較するための方法を紹介してみたいと思います.
はじめにC-statisticsについて簡単に解説したあとに、必要なコマンドを概説したいと思います.
1.C-statiscicsとは
イベント予測モデルあるいは予測スコアの C-統計量は、「イベント発症者と非発
症者を正しく弁別する特性」すなわち判別力(discrimination)を測る指標として知られ
ています.
モデルの判別力(discrimination)といえば、ロジスティック回帰分析を行った後にReceiver Operating Characteristic (ROC) curveを描いて曲線下面積(Area Under Curve, AUC)を求める、というのが一般的なイメージだと思います.これはStataで簡単に行うことができます.これについては別の記事①、記事②を参照してください.
ロジスティック回帰なので、当然時間的な要素は加味されておりません.そこで生存時間分析ではどうか、というと、Harrell’s C concordance statisticと呼ばれる指標でC統計量を算出可能です.解釈としてはAUCと同じように考えればよいようです.すなわち極論かもしれませんが、「1に近ければよいモデル」ということになります.ちなみにこの”C”はconcordanceから来ている、という話です.ちなみにSomers’ Dという統計量もあり、こちらもconcordanceを評価する指標です.Stataでは両方とも算出可能です.
2.StataでのC-statiscicsの算出
StataではCox回帰をしたあとに、.estat concordanceと唱えれば一瞬で結果を得ることができます.これで終わりならいいのですが、異なるモデル同士の比較をしたい場合にちょっと困るわけです.ここで多くの人が挫折してしまっているのではないでしょうか?(including yours truly…)
ここで、サンプルデータを使わせて貰いましょう.drugtrというデータセットはすでにstsetされているデータセットなのですが、こちらを使用します.Cox回帰からのestat concordanceで一気に結果まで出してみます.
use http://www.stata-press.com/data/r11/drugtr, clear
stset
stcox drug age
estat concordance 
一般的にはC統計量は?と聞かれたら、Harrell’s Cを答えればよいです.すなわち0.8086となるわけですね.
ここで問題なのは、①信頼区間がない、②他のモデルと比較できない、です.
Stataでそれを実行しようと思うと、”somersd” というコマンドが必要です. いつものようにfindit somersdとしていただき、snp15_7とかでダウンロードすればいけると思います.
しかしそれだけではない、別の落とし穴が待っています.
- stcoxコマンドを走らせた後に、”predict” コマンドを使ってpositive prediction scoreを出す必要がある. (defaultでは negative prediction score になってしまうため)
- stcoxのデフォルトのcensorship statusは、somersdのcensorship statusと異なる.
- estat concordance のcensor timeがsomersdのものと異なる.
つまり、predictでpositive prediction scoreを出すこと、censorship statusを変更すること、censor timeを変更することが求められます.
estat concordanceで算出した上記の結果を、predictとsomersdを使って再現してみたいと思います.
use http://www.stata-press.com/data/r11/drugtr, clear
stset
stcox drug age
predict hr
/* predictコマンドで推定値を算出 */
generate invhr=1/hr /* 推定値をinverseにする */
generate censind=1-_d if _st==1
/* censorship statusの変更 */
somersd _t invhr if _st==1, cenind(censind) tdist transf(c) /* somersdコマンドを走らせる */ somersdは、上記のようにオプションでtransf(c)とするとHarrell’s C concordance statisticsが算出できます.
ここのsyntaxは理屈抜きでこういうものだと覚えてしまった方が早そうです.
さて、結果ですが、
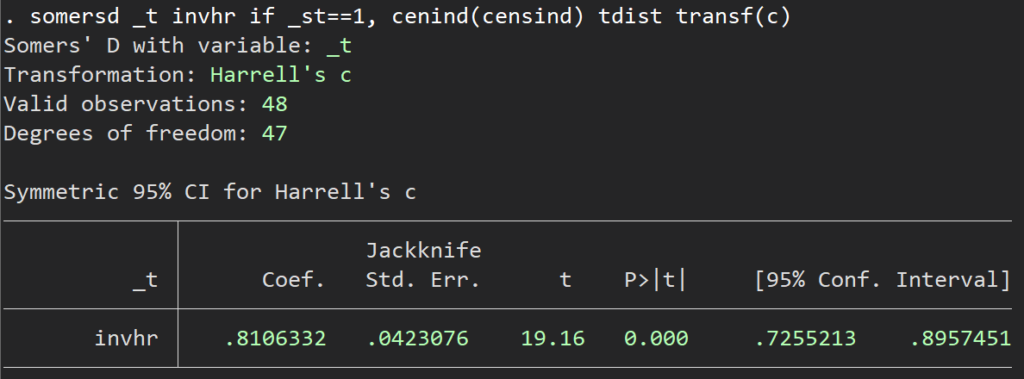
??ちょっと値が違いますね.そうなのです.estat concordanceのHarrell’s Cのところの式をじっと見ていただくと、
Harrell’s C = (E + T/2) / P = 0.8086
となっていますよね?つまり時間のところを少し細工してやらないといけないんです.面倒ですね.
use http://www.stata-press.com/data/r11/drugtr, clear
generate studytime2=studytime+0.5*(died==0)
/* イベントなしの時間を0.5だけ延ばす */
stset studytime2, failure(died)
/* 新たな時間でstsetし直し */
stcox drug age
predict hr
/* predictコマンドで推定値を算出 */
generate invhr=1/hr /* 推定値をinverseにする */
generate censind=1-_d if _st==1
/* censorship statusの変更 */
somersd _t invhr if _st==1, cenind(censind) tdist transf(c) /* somersdコマンドを走らせる */ 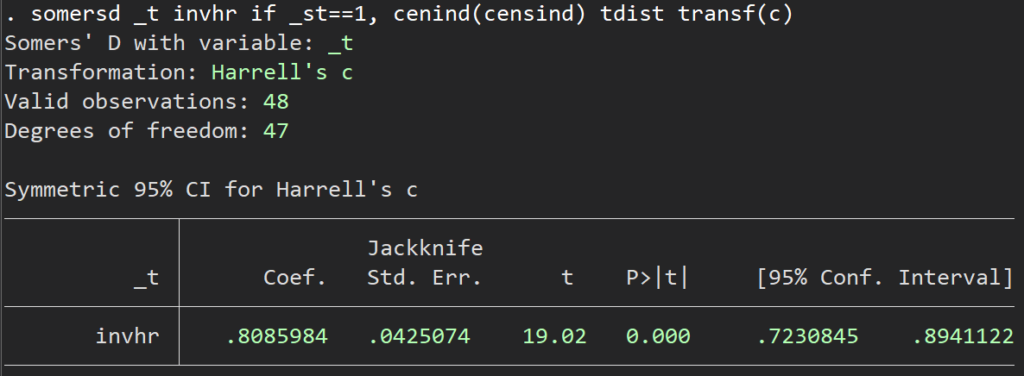
これでC統計量としては同じ値(0.8086)が、しかも95%信頼区間付きで、得られました.
3.他のモデルとの比較
つぎに今日のメインですが、複数のモデルを作成した際にそれを比較する方法についてです.ここでは複数のCox回帰を行ってそれぞれの推定値をもとにC統計量を算出し、最後にlincomで比較する、という流れになります.
use http://www.stata-press.com/data/r11/drugtr, clear
generate studytime2=studytime+0.5*(died==0)
/* イベントなしの時間を0.5だけ延ばす */
stset studytime2, failure(died)
/* 新たな時間でstsetし直し */
*** model 1
stcox drug age
predict hr1 /* predictコマンドで推定値を算出 */
generate invhr1=1/hr1 /* 推定値をinverseにする */
*** model 2
stcox drug
predict hr2
generate invhr2=1/hr2
*** model 3
stcox age
predict hr3
generate invhr3=1/hr3
generate censind=1-_d if _st==1
/* censorship statusの変更 */
somersd _t invhr1 invhr2 invhr3 if _st==1, cenind(censind) tdist transf(c) /* somersdコマンドを走らせる */
*** それぞれのモデルを比較
lincom invhr1-invhr2
lincom invhr1-invhr3
lincom invhr2-invhr3 somersdのsyntaxとしては、時間、それぞれのinverseした推定量を並べていくだけであとの部分は一緒です.lincomeは互いに引き算をすればよいです.大きい方から小さい方に引き算すれば正の値が得られます.
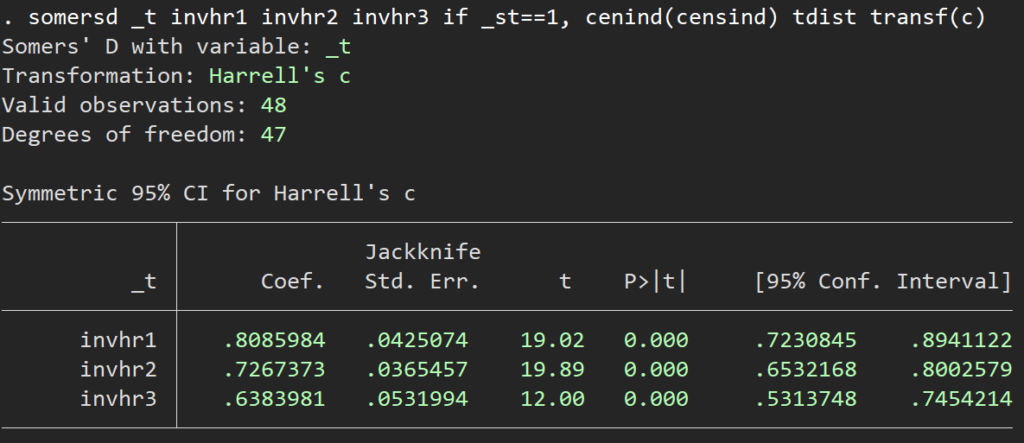
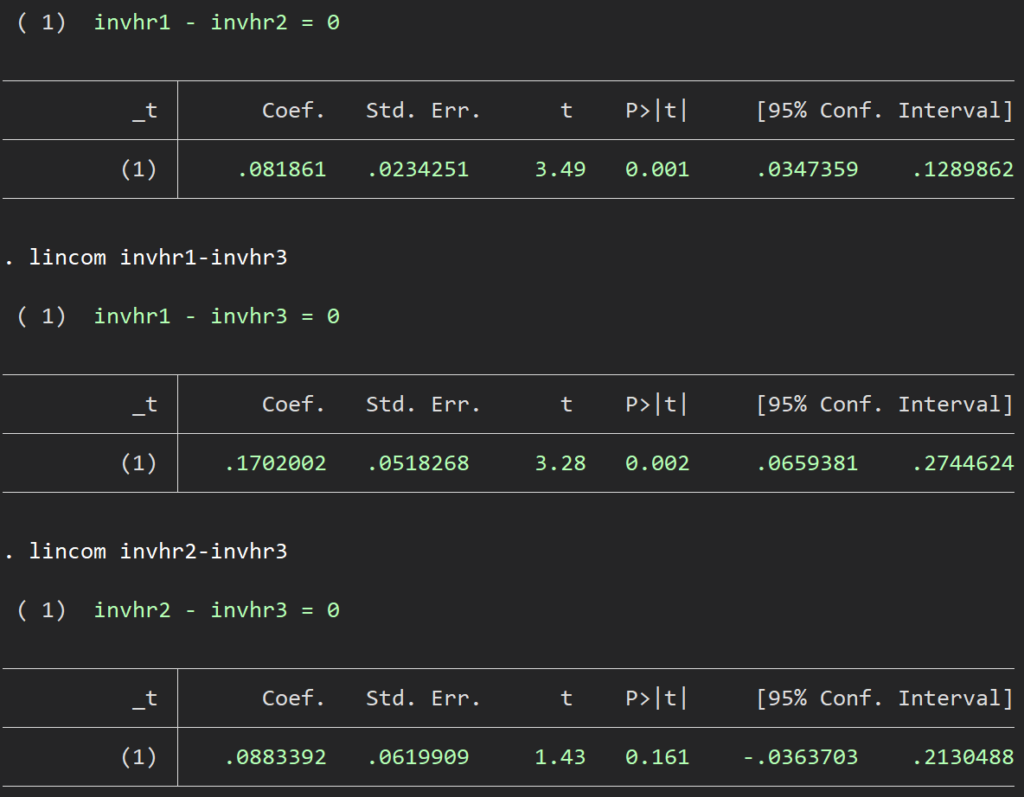
一番上のモデルが最もよく、モデル2と比較すると0.082よく、モデル3と比較すると0.17よい、ということになります.有意差もあります.
まとめ
いかがだったでしょうか?生存時間分析のあとにC-statiscicsを出し、複数のモデルで比較を行うという流れについて説明しました.結構使い勝手がよいと思いますので、これからどんどん使っていこうと思います.


コメント
これで2週間苦しんでいました(泣)
早く知りたかったです。