このブログでは、統計解析ソフトStataのプログラミングのTipsや便利コマンドを紹介しています.
Facebook groupでは、ちょっとした疑問や気づいたことなどを共有して貰うフォーラムになっています. ブログと合わせて個人の学習に役立てて貰えれば幸いです.
以前の記事で、net reclassification improvement (NRI) やintegrated discrimination improvement (IDI)をStataでカスタマイズするための方法を紹介しましたが、今回はその続編です.
1.”incrisk”を入手
この”incrisk”ですが、標準コマンドではないので、別のソースから取りにいかねばなりません.しかし、help incriskとやってもでてきません.これはあるWebサイトに行かねばとれないのです.
自分もこんなコマンドの存在は全く知りませんでした.このコマンドはFacebook groupである方が情報提供をしていただきました.この場を借りてお礼申し上げます.
まずはこちらのWebサイトに飛んでみてください.
Dr. Gary Longton (Fred Hutchinson Cancer Research Center Seattle, WA)
Dr. Margaret Pepe (Fred Hutchinson Cancer Research Center and University of Washington
Seattle, WA)
のお二人が開発してくださった夢のようなコマンドです.以下の一文をコマンドウィンドウに入力してください.
net from https://research.fhcrc.org/content/dam/stripe/diagnostic-biomarkers-statistical-center/files/stata/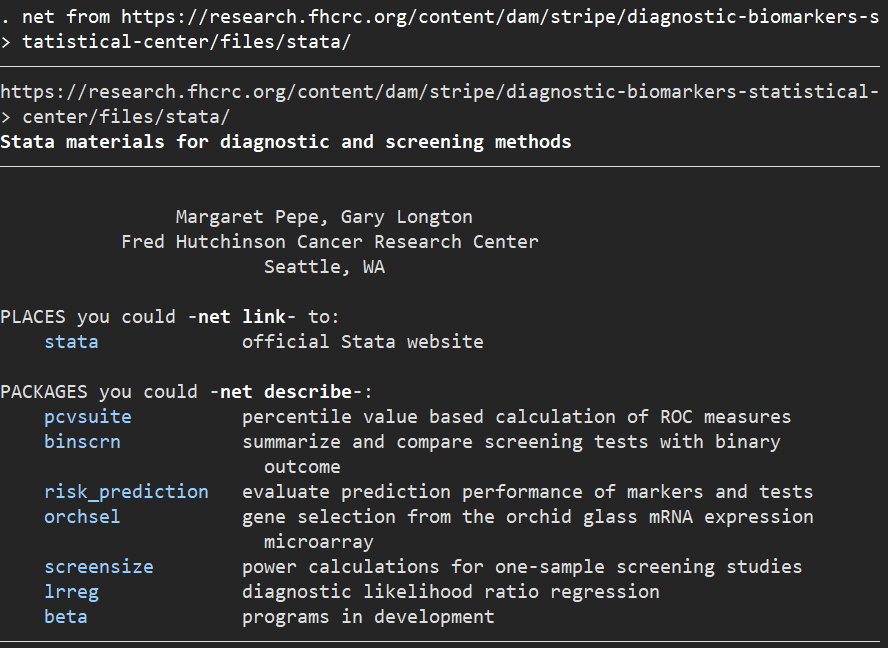
このうちの”risk_prediction”というところをクリックしていただくと、
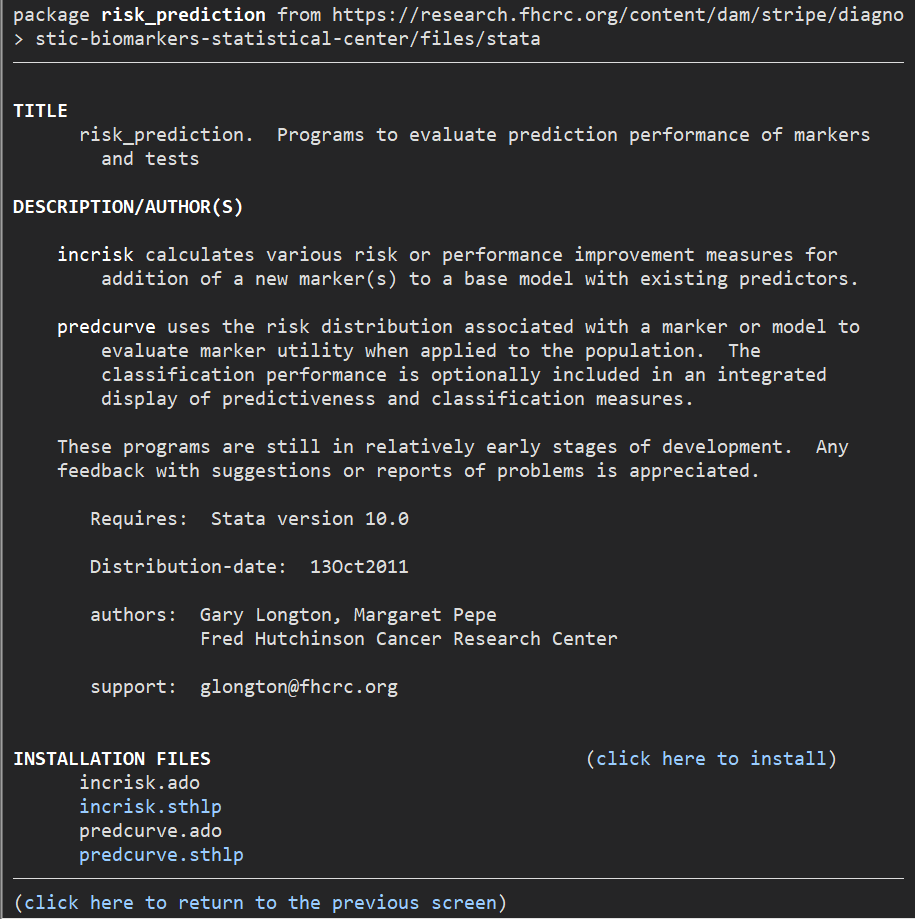
で、いつものようにclick here to install のところをクリックしてもらえばインストール開始となります.
2.どんな結果が返ってくるのか
Helpには以下の結果が返ってくると書いてあります.
- Net Reclassification Improvement (NRI)
- Integrated Discrimination Improvement (IDI)
- changes in each of the Mean Risk Difference (MRD)
- Lorenz measure (L and L^(-1))
- Above Average Risk Difference (AARD)
- standardized Total Gain (TG)
- Area under the ROC curve (AUC)
ここにはかいてありませんけど、reclassification table, ロジスティック回帰のあとのHosmer-Lemeshow testの結果なんかも返してくれます.
これ一つで何度でもおいしいですね.ただし、scalar、macroの類は一切仕掛けられていないので、カスタマイズしたい人向けではありませんね.
それでも十分利用価値があると思います.
3.使い方
使い方は極めて単純です.以下のsyntaxなわけですが、disease_varのところにアウトカム変数であるイベントや病名などを入れていただき、既存の因子をx、新しい因子をyに入れるだけです.
incrisk disease_var [if] [in], x(varlist) y(varlist) [options]x(varlist) base model predictor variables:ベースモデルで既存の因子.複数可.
y(varlist) additional marker or predictor variables:新規マーカー.これも複数可で使いやすくなっています.
デフォルトで10-fold cross-validation + bootstrap (1000 samples)となっていますので、あとは必要に応じてオプションで調整すればオッケーです.よく言われているのがBootstrap法では1000~2000のサンプリングを行うと安定します、ということなのでこのままでもいいんでしょうけど.
とにかく信頼区間はこのbootstrap法で算出しているので、前回紹介した方法とは異なります.
さらにP値は算出されませんのでご注意ください.
なお、bootstrap法を用いる場合は、ランダムに選ばれるため結果が一定しません.そこで必ずSeedを設定しましょう.プログラムの前にset seed ~ とおいて、同時にプログラムを実行してください.
set seed 210121
incrisk outcome , x(covariates) y(newvar1 newvar2) nocvちなみにここでのオプションのnocvはcross-validationを省いたものになります.
まとめ
incriskでいろいろな解析結果を一気に出すことができます.
10-fold cross-validation + bootstrapの結果を基準にしているので、条件設定を色々変えながらほしい結果をゲットしましょう.



コメント