このブログでは、統計解析ソフトStataのプログラミングのTipsや便利コマンドを紹介しています.
Facebook groupでは、ちょっとした疑問や気づいたことなどを共有して貰うフォーラムになっています. ブログと合わせて個人の学習に役立てて貰えれば幸いです.
以前も紹介した、たくさんの変数の変数名やラベルを効率よく変換するための方法を、日常的に扱うデータ加工作業にどのように落とし込むのか、というところに特化して説明してみたいと思います.
例えば、電子カルテのデータを一括で抽出してくるような研究を想定してみましょう.
患者ID、検査実施日付、(場合によっては時間も)、検査項目、検査結果、単位…
といった感じで結果が並んだものが、縦持ちのデータとして、つまり患者一人につき複数のデータが大量に出てくるようなデータセットをイメージしてみましょう.
データの一括抽出をするときには、研究対象者のIDを限定し、データを抜いてくる期間を指定して、その条件を満たす検査結果をすべて抜いてくる、というようなことが多々あります.
必要な項目だけでなく、かなり特殊な検査項目なんかもあったりするので、そういったものは除いて、必要なものだけに限定してデータを整えたりするのですが、それでもかなりたくさんの項目にわたります.
rename 検査項目1 Cr
rename 検査項目2 BUN
...といった感じですべての項目の変数名を調べて変換して、さらに必要に応じて変数ラベルを付けたり、ということをやるわけですが、研究毎に必要なデータ項目が違ってたりします.
自分なりに作業フローをまとめてみたので、もしこういった業務に携わっている方でお役に立てればと思います.
1.作業フロー
まず、作業内容の全体像を俯瞰してみます.一括抽出した縦持ちの検査データを、項目をずらっと横に並べた横持ちデータにすることが目標です.
まず出来上がりのデータセットのイメージから.
- 同じ対象者で同日に測定した項目をずらっと横に並べたい場合
⇒ IDと検体採取日をキー情報として、項目名を順序情報にしてreshape wideします. - 同じ対象者で、別々の日付に測定したものでもいいので、とにかく欠測のないデータを横に並べたい場合
⇒ IDをキー情報として、項目名を順序情報にしてreshape wideしますが、このとき測定日情報も一緒に並べ替えます.
こんな感じのイメージです.
| ID | 採取日 | WBC | Hb | Plt | BUN | Cr | UA |
| ID | WBC 採取日 | WBC 結果値 | Hb 採取日 | Hb 結果値 | Plt 採取日 | Plt 結果値 | BUN 採取日 | BUN 結果値 | Cr 採取日 | Cr 結果値 | UA 採取日 | UA 結果日 |
スタート地点として、受け取ったデータはこんな感じになっていることでしょう.
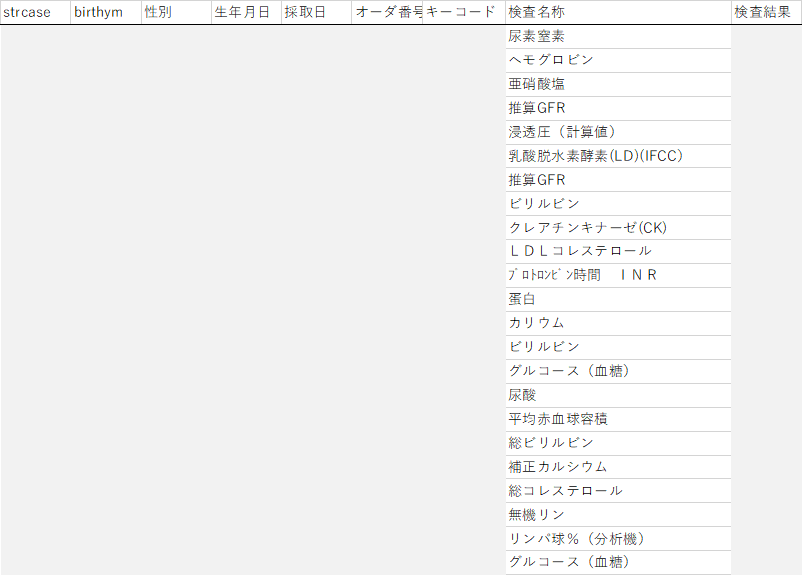
ちょっとボカシをいれていますが、実際のデータセットから抜き出してきました.
よく見ればスペースとか、()とか入ってしまっていますし、ハイフンとか入ってしまっていることもあります.
こういった文字列は変数名としては適切ではありませんので、1つずつ手で修正を加えなければならないのです.
扱うデータセットが多岐にわたっている、それも異なる診療科からデータを要求される、みたいな状況になっていると、ある程度作業は自動化しておきたくなります.
(もちろん縦持ちのデータのまま渡したりもするんですが、これもらっても加工できませんけどぉ、、、という反応が高い確率で返ってきますので、結局加工まで手伝ってしまいます.)
変数の定義書を作ったりするのも同じくらいの手間なので、どうせなら一緒にやってしまうか、という感じです.
さて、そんな作業を研究者自ら手元で行えるようになれば作業軽減となるばかりでなく、他の研究にも研究者自身が取り組むことができるようになりますので、ぜひここで書かれている内容を咀嚼していただいて、身に着けていただけるとよいのではないかと思います.
さて、作業フローですが、
- 一括抽出したデータセットをStataで読み込む(必要に応じて匿名化など)
- 検査名称または検査項目のローカルコードで重複削除する(項目名のリストが完成)
- 一旦エクセルなどにエクスポート
- 必要な検査項目を選択し、それ以外はリストから削除する(±変数名を手作業でエクセルシートに書き込む) ⇒ 別名で保存する
- 別名で保存した変数リストをStataで再び読み込む
- リストを基にして、命令をDo fileに書き込むためのプログラムを作成する(以降はコチラ参照)
- データセットの加工、変数名、ラベルの設定を
ということで、ステップごとに解説してみたいと思います.
2.作業工程1~3
ここは基本的なコマンドで作業が終わります.しかしデータ自体は大きいと思いますので、思いのほか時間がかかると思います.(先日、1億件以上のデータを初めて展開したのですが、さすがに10分以上かかりました.)
import excel "データセット名称", sheet("シート名") firstrow clear
duplicates drop 検査名称 , force
keep 検査ローカルコード 検査名称 結果
export excel "labmaster", firstrow(variables) replaceこんな感じでしょうか.
出ました、ラブマスター.恋愛の達人!みたいだなぁと思ってニヤニヤしてました.不気味なおっさんですよね.
まあそれはいいとして、この後どうするかというと、エクスポートしたエクセルを手作業で加工するという作業段階に入ります.
3.作業工程4~6
ここで、なぜいちいちエクセルでエクスポートしたのか、疑問に感じられた方もいるかと思います.
それは、手作業での作業においてはやはりエクセルの方がやりやすい、というのが理由です.
いらない変数を行削除で消去したり、変数名の横にべた書きで変数名を新しく入れたりとなかなかに重宝します.
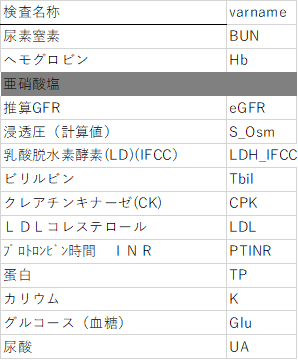
先ほどの表を加工して、右側に変数名を書き込みます.キーとする情報として検査名称または検査ローカルコード(標準コードならJLAC10あたりでしょうか)などを使って元のデータに戻してあげればいいわけです.例えばlocalcodeという変数があったとして、それが検査項目と1:1で対応しているのであれば、次のように書けば変数名とラベル名を設定することができるでしょう.
preserve
import excel "${wd}labmaster2.xlsx", firstrow allstring clear
list localcode 検査名称 varname
tempname fh
local N = _N
file open `fh' using my_labels.do , write replace
forvalues i = 1/`N' {
file write `fh' "replace 検査名称 ="
file write `fh' `""`= varname[`i']'""'
file write `fh' " if 検査名称=="
file write `fh' `""`= 検査名称[`i']'""' _newline
}
file close `fh'
import excel "${wd}labmaster2.xlsx", firstrow allstring clear
list localcode 検査名称 varname
tempname fh
local N = _N
file open `fh' using my_labels2.do , write replace
forvalues i = 1/`N' {
file write `fh' "capture label variable `= varname[`i']' "
file write `fh' `""`= 検査名称[`i']'""' _newline
}
file close `fh'
restore
** Give variables label
do my_labels.do
…検査項目名で入っているのを、varnameで入っている変数名に置き換えてね!という命令文をdo fileに次々に書き込んでいく感じになります.
ここでどんな感じのdo fileが出来上がるかをお見せします.ある研究のために作ったプログラムをチラ見せしてしまいます.このような命令文すらプログラムで自動的に生産することができるわけです.
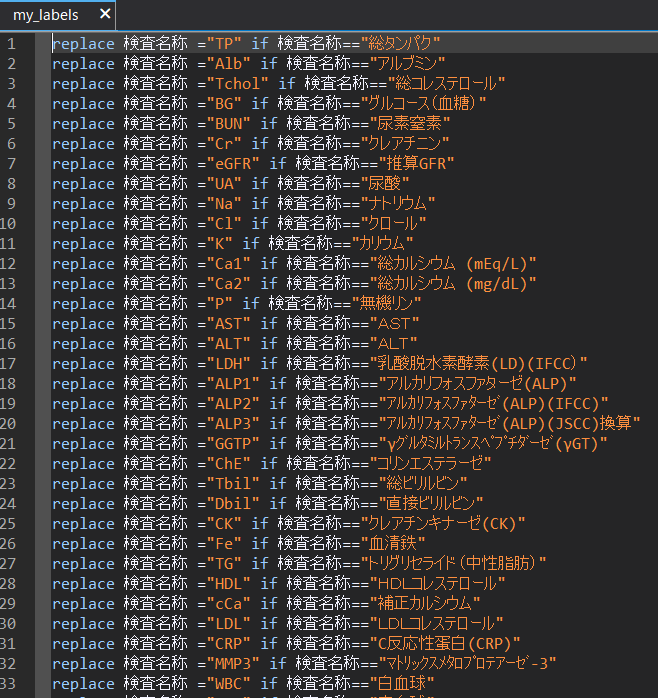
do fileを作り出すためのプログラムの設定方法を理解するのはなかなか大変ですが、色々試してみて、出来上がったdo fileを開いて微調整をしてみるのがいいでしょう.それでやり方がだんだんわかってくると思います.
4.データ加工の最終段階
さて、ここまではデータ加工用のdo fileをせっせと作ってきたわけですが、それはどんなことをやらせようとしているのでしょうか?
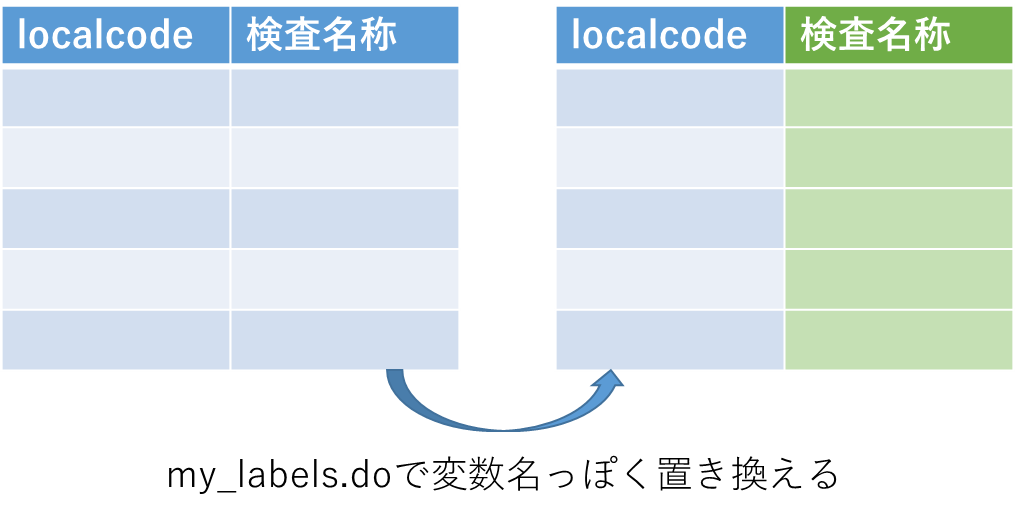
その変数名っぽい文字列を実際の変数名として活用します。具体的にはデータを横持ちにして、それぞれの変数名を割り当てるという作業をこれから行っていくという理解をしてください.
検査結果が文字列のものを「結果」、数値のものを「結果数値」という風にして格納している場合を想定していただきますと、
encode localcode, gen(code)
reshape wide localcode varname 結果 結果数値 単位, i(ID 採取日時) j(code)
forvalues n = 1/20 {
gsort -結果数値`n'
if 結果数値`n'~=. {
local v = 検査名称`n'[1]
rename 結果数値`n' `v'
}
else {
local v = 検査名称`n'[1]
rename 結果`n' `v'
}
}
do my_labels2.doこんな風になるかと思います.まず、localcodeが文字列の場合には encode して数値データの変数を作ります.それぞれの検査項目に数字が割り当てられます.これをreshapeコマンドのj( )の中にいれることで、項目が順番に並び、ID+採血実施日の2つのキー情報を使って横持ちデータに加工します.
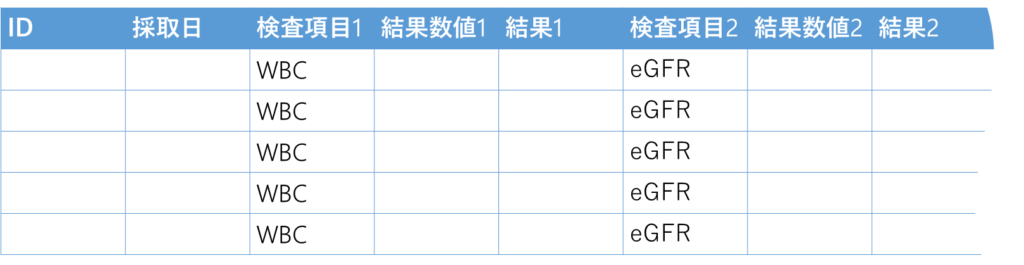
こんな感じで横持ちになりますが、WBCもeGFRも数値データですので「結果数値#」という変数に格納されている結果だけがあればいいわけで、これの変数名をそれぞれWBCとかeGFRに直せればいいわけです.
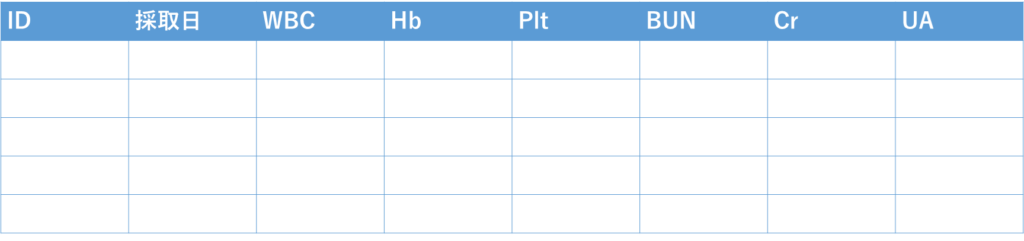
この操作を上のプログラムでは実行していると思っていただければと思います.
5.まとめ
いかがでしたでしょうか?
なかなか複雑そうに見えるかもしれませんが、実際には一度作ってしまえばあとは使いまわしができますので大変重宝しています.少しでも手数を減らす工夫をするのがプログラミングの醍醐味ですよね.



コメント