このブログでは、統計解析ソフトStataのプログラミングのTipsや便利コマンドを紹介しています.
Facebook groupでは、ちょっとした疑問や気づいたことなどを共有して貰うフォーラムになっています. ブログと合わせて個人の学習に役立てて貰えれば幸いです.
さて、以前の記事で、eGFRの年間低下速度について説明しましたが、実際にどうやるのか、というところにまで踏み込んでいませんでした.
確かにこの計算方法についてはあまり詳しく載っている資料が乏しく、自分自身も手探りでやっているのが現状ですので、もし確からしい情報を入手された方がいたらぜひ情報共有をお願いします.
1.実際のSlope計算方法
mixedのコマンドのあとに結果をpredictすることでランダムスロープとランダム切片を取得します。その方法はBLUPという手法をとったとJASN2019の論文には書いてあります。
as a primary exposure, we used linear mixed models with an unstructured variance-covariance matrix, random intercept, and random slope for each individual to estimate slope (mixed model slope)12.
ここで引用されている文献がBLUPという方法に触れています.
また、StataCorpのGutiérrezらが書いたまとめの中には以下のように書かれています.
Random effects are not estimated, but they can be predicted (BLUPs).
また、このまとめの中では、mixed effect modelを走らせたあとに
predict r1 r0, reffect
と入力することでr1、r0にそれぞれランダム効果を出してくれることになっています.
最終的には固定効果を_b[_cons], _b[変数名]として出力し、r0とr1とそれぞれ足し算することで固定効果ならびに変量効果の両方を加味した傾きを求めることができるわけです.
OutcomeをeGFR、visit番号をvisit、IDをcase_idとすると、以下のようにして求めることになるかと思います.visitは月に1回あるとすると、visitが一つ増えるごとに1か月経過することになりますので、年間変化率を出そうとすると12倍する必要があります.
mixed eGFR visit || case_id:visit, cov(unstructured)
gen fixslope = _b[visit]
gen fixcons = _b[_cons]
predict ranslope rancons, reffect
gen slope_full = (fixslope + ranslope) *12これをvisitの代わりにbaselineからの経過日数とするなら最終的には日にち単位となるため、365倍することになります.
2.Visitの回数や間隔が与える影響
リアルワールドの診療データを使った研究などを行う場合には、コホート研究と異なり、Crを測定したタイミングそのものに意味があります.
つまりどういうことかというと、1~3か月おきに定期受診する患者さんが多いと思いますが、その受診間隔はどうやって決めているでしょうか?ランダムですか?
多くの場合には、その患者さんの状態が影響しているはずです.
また、薬を新たに処方したときや、患者さん自身が調子悪くなって受診したりした場合にはいつもより短い間隔で受診してくるかもしれません.また、入院すると採血の頻度が増えますよね.
採血の値がある一定の時期に固まっていると、その値に全体の結果が引っ張られます.通常の外来ではeGFRが60くらいの人が、感染症などを契機にして集中治療室での治療が必要な状態で入院したときの状況を想像してみてください.
その人がeGFR 40程度になってしまった.でも無事回復して退院した.
こんな状況だと下の図のようになるはずです.
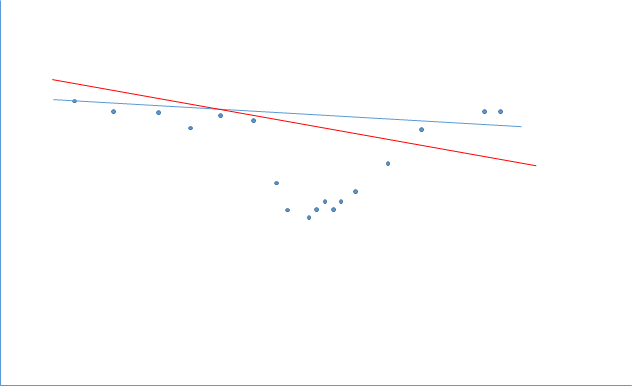
ちょっと腎機能が悪くなった時期を除くと青線のようにSlopeが引けるはずなのが、この値の影響によって赤線のようなSlopeになってしまうのです.
このような影響を排除するためにどのように工夫すればよいでしょうか?残念ながらこれを解消するためのエビデンスは乏しいのが現状です.考えられる対応策としては以下のようなものがあるかもしれません.
- 入院中のデータは使わないことにする
- 1か月に1回ずつのデータにする(最も値の高いもの、あるいは平均値などの代表値に置き換える)
いずれにしても定まった定義は今のところないですので、研究者自らが定義づけを行う必要があります.
3.月ごとのデータに集約する方法の一例
さて、データ加工の工夫として、ベースラインからの日数でVisit番号を新たに付与する方法を考えてみたいと思います.
検査をした日とベースライン日の差をgapとして、30日ごとに句切れ目を入れてVisit番号を振る方法です.
sort case_id gap
bysort case_id: gen visit = irecode(gap, 0, 30, 60 , 90, 120, 150, 180, 210, 240, 270, 300, 330, 360, 390, 420, 450, 480, 510, 540, 570, 600, 630, 660, 690, 720)これだと1年360日計算になってしまうので、少しずつずれて行ってしまいますので、観察期間が長くなっていくようでしたらもう少し日付の割り振りの仕方には工夫が必要かもしれません.
そして、同じvisit番号を持つレコードは一定の基準で一つに統一します.(最大値もしくは平均値など)
平均値を同じvisitごとに出したいのであれば、
sort case_id visit gap
bysort case_id visit : egen eGFR_vave = mean( eGFR )このようにやれば同じvisitの値の平均値をとることができます.
まとめ
Mixed effects modelを用いたeGFR slope推定方法について解説しました.しかしまだReal-world dataを用いたSlope計算についてはエビデンスが不足していると思いますので、引き続きcatch upしていきます.
最新の情報が入手できたらまた情報共有をさせていただきます.



コメント